Tabla de Contenidos
5. Pandas
Pandas es una librería en cierto sentido similar a NumPy. Pero si NumPy únicamente contiene vectores, matrices, tensores ,etc junto con operaciones matemáticas. Con pandas tenemos mas cosas como nombrar a las columnas con un nombre , incluir un índices o generación de gráficas.
Mas información:
Importación
- Importar pandas
import pandas as pd
DataFrames
- Crear un DataFrame a partir de la matriz de datos y el nombre de las columnas con
DataFrame
tipo=['SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD'] capacidad=[0.5, 0.5, 0.24, 0.48, 1, 0.512, 1, 0.48, 0.12, 0.96, 0.256, 0.512, 1, 2, 0.5, 2, 2, 1, 1.5, 4, 2, 4, 6, 5, 8, 10, 12, 14, 16, 18] precio=[101, 51, 27, 44, 86, 101, 138, 50, 22, 83, 41, 78, 126, 183, 91, 48, 51, 37, 55, 81, 48, 88, 187, 146, 240, 360, 387, 443, 516, 612] data=zip(tipo,capacidad,precio) columns=['tipo', 'capacidad','precio'] df=pd.DataFrame(data, columns=columns)
zip es similar a np.column_stack de numpy pero ha usado zip en vez de column_stack ya que hay datos de tipos string. Si todos los datos hubieran sido números de podría haber usado column_stack
- Añadir una nueva fila al DataFrame con
append.
df=df.append({'tipo':"SSD","capacidad":1,"precio":214},ignore_index=True)
Mas información:
Acceso a disco
- Guardar los datos de un DataFrame
df.to_csv("datos.csv", index=False)
index=False ya que sino creará en disco una columna extra con el nº de la fila a modo de índice
tipo,capacidad,precio SSD,0.5,101 SSD,0.5,51 SSD,0.24,27 SSD,0.48,44 ........ HDD,14.0,443 HDD,16.0,516 HDD,18.0,612 SSD,1.0,214
- Cargar los datos desde un fichero de texto llamado "datos.csv" y cuyo separador es una coma.
df=pd.read_csv("datos.csv",sep=",")
index=False ,al leer el fichero se deberá añadir el parámetro index_col=0
df.to_csv("datos.csv")
df=pd.read_csv("datos.csv",index_col=0)
- Cargar los datos desde una base de datos relacional
import sqlalchemy
connection = sqlalchemy.create_engine('mysql+pymysql://mi_usuario:mi_contrasenya@localhost:3306/mi_database')
df=pd.read_sql("SELECT * FROM mi_tabla",con=connection)
Crear un DataFrame desde una base de datos relacional es tan sencillo como crear la conexión con sqlalchemy.create_engine y luego con pandas llamar a read_sql.
conda install -c anaconda sqlalchemy
Información
- Información general del DataFrame
df.info()
<class 'pandas.core.frame.DataFrame'> RangeIndex: 31 entries, 0 to 30 Data columns (total 3 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 tipo 31 non-null object 1 capacidad 31 non-null float64 2 precio 31 non-null int64 dtypes: float64(1), int64(1), object(1) memory usage: 872.0+ bytes
- Dimensión de los datos (filas y columnas)
df.shape
(31, 3)
- Mostrar las primeras filas
df.head()
tipo capacidad precio 0 SSD 0.50 101 1 SSD 0.50 51 2 SSD 0.24 27 3 SSD 0.48 44 4 SSD 1.00 86
- Mostrar las últimas filas
df.tail()
tipo capacidad precio 26 HDD 12.0 387 27 HDD 14.0 443 28 HDD 16.0 516 29 HDD 18.0 612 30 SSD 1.0 214
Estadística
- Estadística descriptiva
df.describe()
capacidad precio
count 28.000000 28.000000
mean 4.092143 157.428571
std 5.209273 158.877178
min 0.120000 22.000000
25% 0.512000 50.750000
50% 1.750000 89.500000
75% 5.250000 184.000000
max 18.000000 612.000000
- Media de una columna
df.precio.mean()
157.42
- Desviación estándar de una columna
df.precio.std()
158.87
- Suma de una columna
df.precio.sum()
4408.0
- Máximo de una columna
df.precio.max()
612.0
- Mínimo de una columna
df.precio.min()
22.0
- Correlación entre columnas
df.corr()
capacidad precio
capacidad 1.000000 0.962542
precio 0.962542 1.000000
Acceder a los datos
Lo normal es que queramos acceder a los datos siempre por columnas. Así que explicaremos únicamente esa forma.
- Acceder a una columna.
df['capacidad'] df.loc[:,'capacidad'] df.capacidad
loc hay que indicar que queremos todas las filas para ello usamos el :
- Acceder a varias columnas
df[['precio','capacidad']] df.loc[:,['precio','capacidad']]
- Acceder a todas las columnas excepto a una concreta (se usa para no acceder a la columna del resultado)
df.loc[:,df.columns!='tipo']
- Acceder a todas las columnas
df df[:] df.loc[:,:]
- Acceder a una columna por índice
df.iloc[:,1]
- Acceder a la última columna por índice
df.iloc[:,-1]
- Acceder a varias columnas por índice
df.iloc[:,[0,-1]]
Columnas
- Obtener el nombre de todas las columnas
df.columns
['tipo', 'capacidad', 'precio']
- Obtener el nº de columnas
len(df.columns)
- Obtener el nombre de la columna por el Nº de columna
df.columns[0]
- Cambiar el nombre de una columna con
rename. Se pasa un diccionario cuya clave es el nombre actual y el valor es el nuevo nombre.
#Cambiamos el nombre de la columna "tipo" al nuevo nombre "target"
df.rename(columns={'tipo': 'target'}, inplace=True)
new_df=df.rename(columns={'tipo': 'target'})
- Notar que al indicar
inplace=Truese hace la modificación en el propioDataFramey no hace falta asignarlo a otro nuevoDataFrame - Lo normal es que la columna a predecir la llamemos
target
- Reordenar las columnas con
reindex
#Ahora la columna target está al final df=df.reindex(columns=['capacidad','precio','target'])
- Notar que es necesario asignar el
DataFramea un nuevoDataFrame - Lo normal es que la columna a predecir se ponga al final
- Añadir la nueva columna
velocidadal inicio del DataFrame
datos_nueva_columna=[1000, 1250, 6500, 2500, 2750, 2500, 1000, 1500, 2250, 5500, 2750, 4250, 5000, 3750, 2500, 6500, 5250, 5250, 3250, 3500, 7250, 6250, 2250, 3500, 4250, 6000, 2000, 3000, 5250, 2500] df.insert(0,"velocidad",datos_nueva_columna)
- Añadir la nueva columna
velocidadantes del "precio"
datos_nueva_columna=[1000, 1250, 6500, 2500, 2750, 2500, 1000, 1500, 2250, 5500, 2750, 4250, 5000, 3750, 2500, 6500, 5250, 5250, 3250, 3500, 7250, 6250, 2250, 3500, 4250, 6000, 2000, 3000, 5250, 2500] df.insert(2,"velocidad",datos_nueva_columna)
- Añadir una nueva columna calculada
df.insert(3,"calculada",df.precio*df.capacidad)
tipo capacidad precio calculada 0 SSD 0.50 101 50.50 1 SSD 0.50 51 25.50 2 SSD 0.24 27 6.48 3 SSD 0.48 44 21.12 4 SSD 1.00 86 86.00 ...........
- Modificar una columna
#Pasamos de euros a dolares df.precio=df.precio*1.13
- Borrar la columna llamada
calculada
df.drop(columns = ['calculada'], inplace = True) new_df=df.drop(columns = ['calculada'])
Filtrado
- Filtrar por una condición: Cuyo tipo es "SSD"
df[df.tipo=='SSD']
- Filtrar por dos condiciones: Cuyo tipo es "SSD" y el precio es menor que 100
df[(df.tipo=='SSD') & (df.precio<100)]
DataFrame así que se pueden aplicar todos los métodos de los DataFrame.
- Valores únicos
df.tipo.unique()
array(['SSD', 'HDD'], dtype=object)
Datos inválidos
Vamos ahora a crear un DataFrame con datos inválidos.
tipo=[None, 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD', 'SSD',
'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD', 'HDD']
capacidad=[0.5, math.nan, 0.24, None, 1, 0.512, 1, 0.48, 0.12, 0.96, 0.256, 0.512, 1, 2, 0.5, 2, 2, 1, 1.5, 4, 2, 4, 6, 5, 8, 10, 12, 14, 16, 18]
precio=[101, 51, math.nan, 44, None, 101, 138, 50, 22, 83, 41, 78, 126, 183, 91, 48, 51, 37, 55, 81, 48, 88, 187, 146, 240, 360, 387, 443, 516, 612]
data=zip(tipo,capacidad,precio)
columns=['tipo', 'capacidad','precio']
df=pd.DataFrame(data, columns=columns)
- Obtener el número de datos a
NaNoNonede cada columna
df.isnull().sum()
tipo 1 capacidad 2 precio 2 dtype: int64
- Borrar aquellas filas que tiene el valor
NaNoNone
df=df.dropna() df.isnull().sum()
tipo 0 capacidad 0 precio 0 dtype: int64
Gráficas con Seaborn
Con dataframes lo sencillo usar seaborn
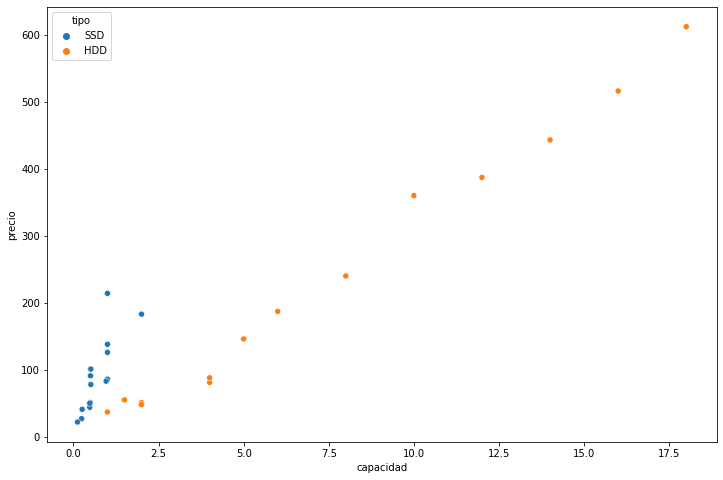
- Scatter plot por el tipo
figure=plt.figure(figsize=(12,8)) axes = figure.add_subplot() sns.scatterplot(x="capacidad", y="precio", hue="tipo",data=df,ax=axes)
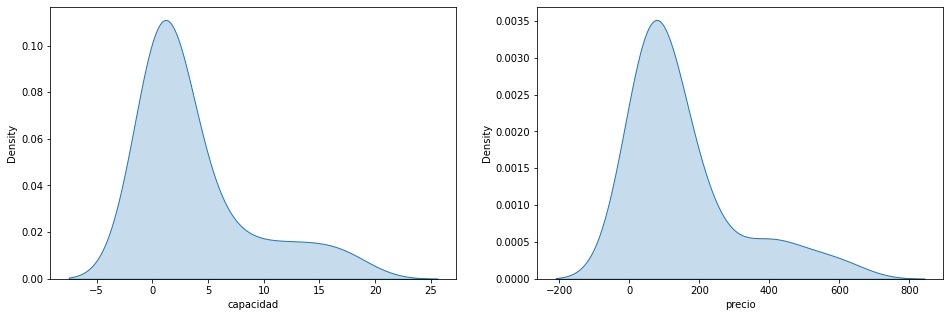
- KDE
figure=plt.figure(figsize=(16,5)) axes = figure.add_subplot(1,2,1) sns.kdeplot(x="capacidad",data=df,fill=True,ax=axes) axes = figure.add_subplot(1,2,2) sns.kdeplot(x="precio",data=df,fill=True,ax=axes)
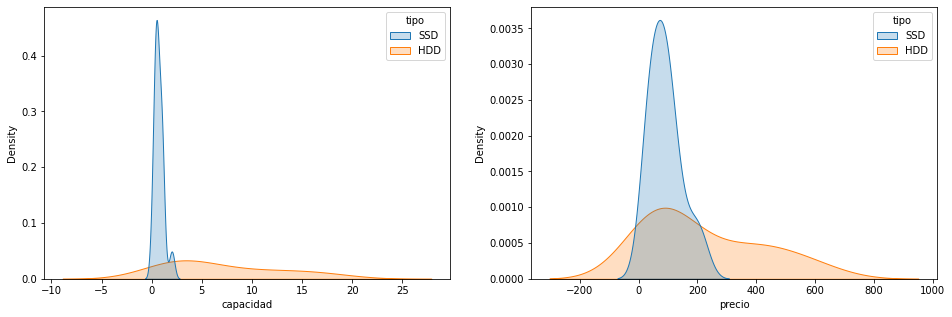
- KDE por el tipo
figure=plt.figure(figsize=(16,5)) axes = figure.add_subplot(1,2,1) sns.kdeplot(x="capacidad",hue="tipo",data=df,fill=True,ax=axes) axes = figure.add_subplot(1,2,2) sns.kdeplot(x="precio",hue="tipo",data=df,fill=True,ax=axes)
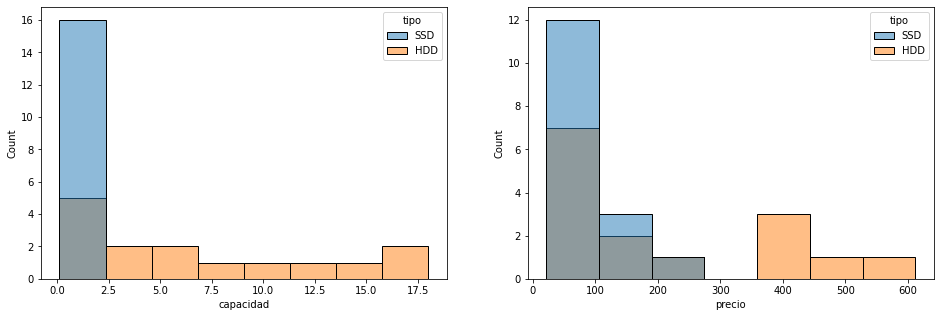
- Histograma por tipo
figure=plt.figure(figsize=(16,5)) axes = figure.add_subplot(1,2,1) sns.histplot(x="capacidad",hue="tipo",data=df,ax=axes) axes = figure.add_subplot(1,2,2) sns.histplot(x="precio",hue="tipo",data=df,ax=axes)
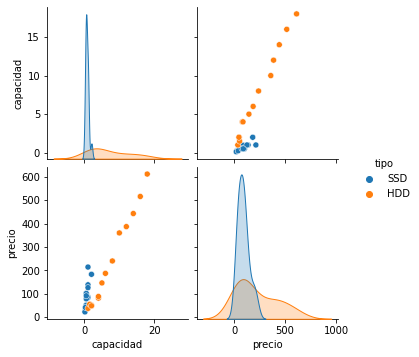
- Pair plot
pairplot=sns.pairplot(df,hue="tipo")
Ejercicios
Ejercicio 1.A
Crea un DataDrame con los datos que proporciona load_iris. Recuerda que la propiedad feature_names retorna los nombres. La columna de los tipos de flor la debes llamar tipo_flor
- Grábalo a disco y mira el fichero resultante.
- Ahora prueba a cargarlo
- Grábalo a disco con otro nombre pero ahora sin el parámetro
index=Falsey mira la diferencia con el anterior fichero.- Ahora prueba a cargarlo
- Muestra el nombre de las columnas y los tipos de datos de cada una de ellas.
- Imprime por pantalla "El nº de número de características es: NNNNN y el número de muestras es: NNNNNN"
- Imprime por pantalla las primeras filas
- Imprime por pantalla las últimas filas
- ¿Cuando ocupa en memoria el DataFrame?
Ejercicio 1.B
Siguiendo con el DataFrame anterior imprime por pantalla:
- El tamaño medio del ancho del pétalo
- La desviación del ancho del pétalo
- El máximo ancho del pétalo
- El mínimo ancho del pétalo
Ejercicio 1.C
Siguiendo con el DataFrame anterior:
- Imprime por pantalla el nombre de las columnas como un array
- Imprime por pantalla el nombre de la primera columna
- Imprime por pantalla el número de columnas que hay.
- Cambia el nombre de la columna "tipo_flor" por el de "target"
- Mueve la columna "target" al final del DataFrame
- Inserta una nueva columna que sea el área del pétalo. Deberás insertarla antes de la última columna.
- Inserta una nueva columna que sea el área del sépalo. Deberás insertarla antes de la última columna.
Ejercicio 1.D
Siguiendo con el DataFrame anterior imprime por pantalla:
- El ancho del pétalo de las flores de tipo "0" pero cuyo largo del pétalo sea entre [1.3,1.9]
- El ancho del sépalo de las flores de tipo "1"
- Indica los tipos de flores que hay.
- Indica cuantos valores de la columna "target" son "null"
- Indicar cuantos valores del DataFrame son "null"
- ¿Como se borrarían aquellas filas que tienen algún datos que es null?
Ejercicio 1.E
Siguiendo con el DataFrame anterior y usando Seaborn:
- Muestra un scatter plot del ancho del pétalo en el eje X y el ancho del sépalo en el eje Y según el tipo de flor.
- Muestra el gráfico KDE de la distribución del largo del pétalo y del largo del sépalo.
- Muestra el gráfico KDE de la distribución del largo del pétalo y del largo del sépalo pero separado por el tipo de flor.
- Muestra un pairplot
Ejercicio 2.A
Descarga el siguiente fichero tiempos_red_neuronal.csv que contiene los segundos que ha tardado en entrenarse una red neuronal según el nº de épocas y la función de activación usada
- Abre el fichero con un editor de texto y comprueba que formato tiene
- Carga el fichero con pandas
- Muestra las columnas que tiene
- Renombra la columna
tallaaepoca - Muestra cuantas filas hay
- Muestra cuantos valores son null o NaN en cada columna
- Muestra el % de valores a null o NaN en cada columna
- Borra las filas que tengan algún valor a null o NaN
- Muestra las estadísticas de cada columna. ¿Podrías borrar alguna? ¿Explica porqué? En caso afirmativo borra la columna
- Indica que funciones de activación se han usado
- Indica hasta cuantas épocas se ha entrenado la red
- Indica la función de activación que ha tenido el mayor tiempo en la última época
Ejercicio 2.B
Siguiendo con el DataFrame anterior:
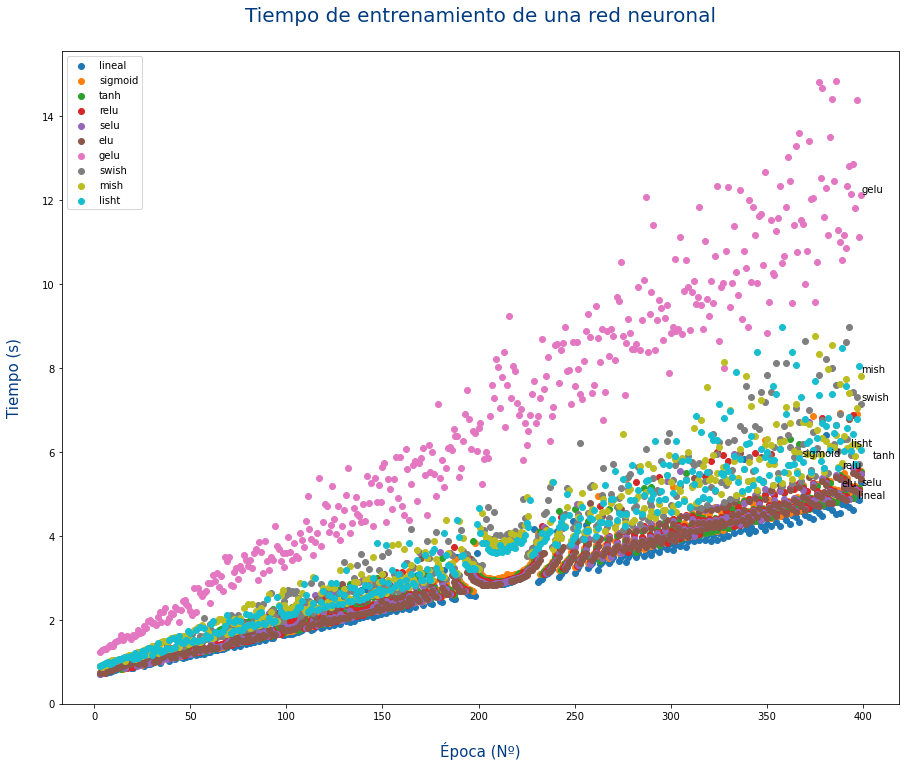
- Muestra un scatter plot
- Eje X:Época
- Eje Y:Tiempo
- Separa los datos por colores según la función de activación
- Añade un título al gráfico y a los 2 ejes.
¿Ves algo raro en los datos?
Ejercicio 2.C
Siguiendo con el DataFrame anterior:
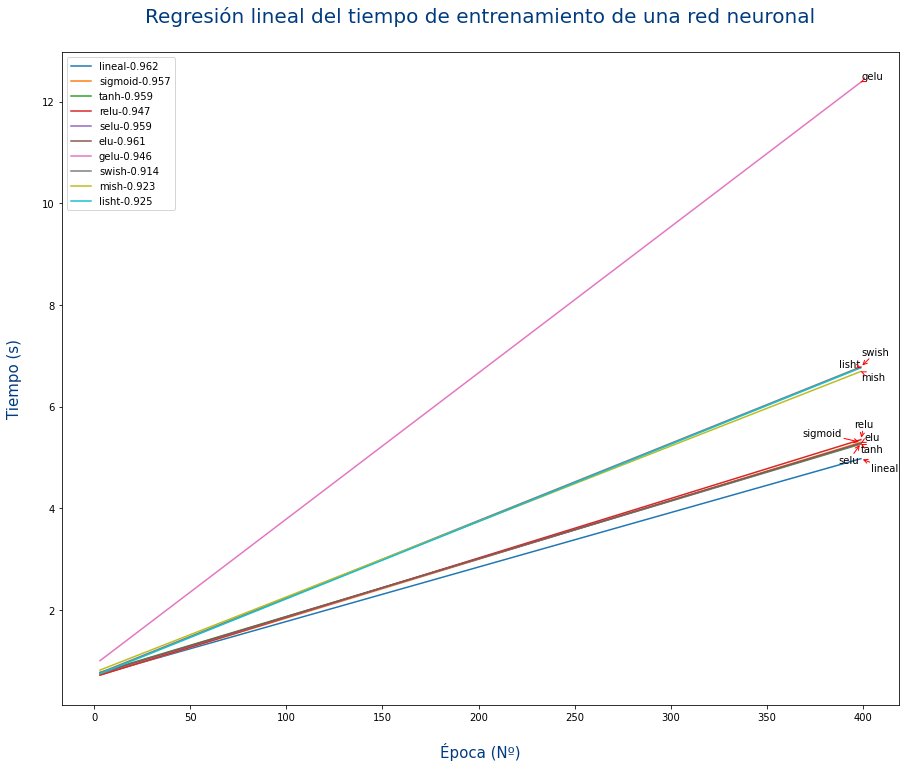
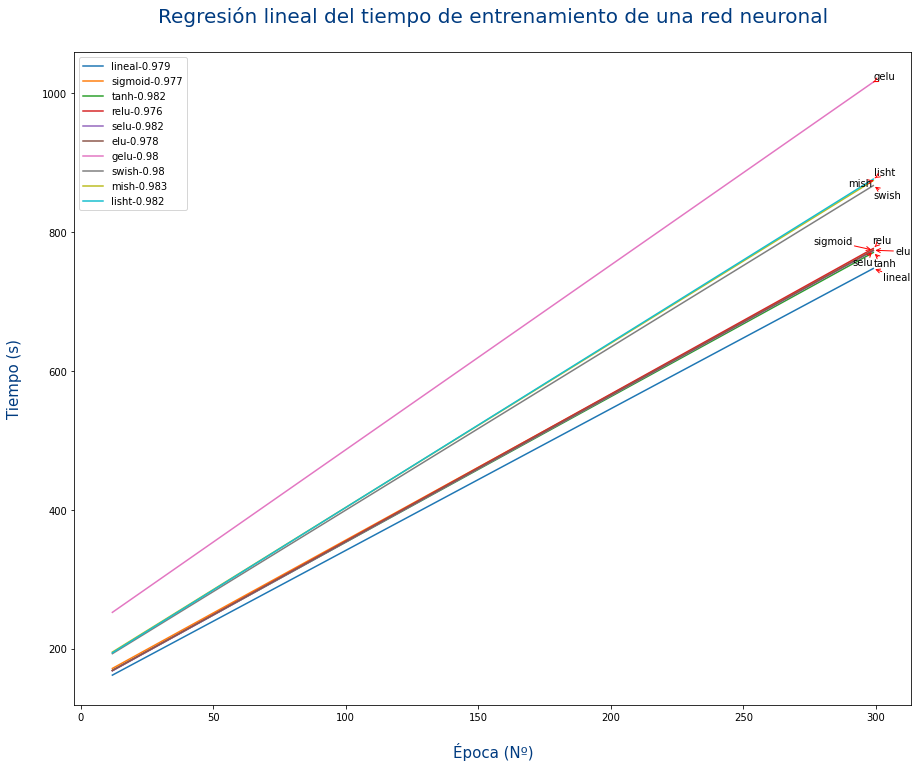
- Haz una regresión lineal de los datos para cada tipo de activación.
- Haz un gráfico que:
- Muestre la recta de la regresión de cada función de activación.
- En la etiqueta de cada función de activación se muestre también el valor de R² para cada uno de ellos.
Ejercicio 2.D
Siguiendo con el DataFrame anterior:
La gráfica no deja claro cda una de las rectas. Para mejorarlo se podría anotar al final de cada recta el nombre de la función de activación tal y como se muestra en el siguiente gráfico:
Para poner texto en una gráfica se usa el método annotate y la librería adjustText - automatic label placement for matplotlib
Ejercicio 3.A
Usando la función plot_metrics muestra las gráficas de las pérdidas por épocas de las siguientes redes neuronales para el DataFrame de las flores:
| Nº Neuronas en cada capa |
|---|
| 2,4,2,1 |
| 4,8,4,1 |
| 8,16,8,1 |
| 4,8,4,2,1 |
| 8,16,8,4,1 |
| 16,32,16,8,1 |
| 32,64,32,8,1 |
| 64,128,64,8,1 |
| 8,16,32,64,32,16,8,1 |
Además:
- Deberás mostrar los 9 subplots en la disposición de 9x1 (9 filas y 1 columna)
- El título de cada subplot será el nº de neuronas por capa
Ejercicio 3.B
Siguiendo con el DataFrame anterior:
- Para las 4 columnas de las características de las flores y el tipo de flor, crea 5 nuevas columnas que se llamen igual pero con el prefijo
normalizado_. Esas 5 nuevas columnas tendrán los valores según la fórmula:
$$ valor = \frac {X-min}{max-min} $$
- Para las 4 columnas de las características de las flores y el tipo de flor, crea 5 nuevas columnas que se llamen igual pero con el prefijo
standarizado_. Esas 5 nuevas columnas tendrán los valores según la fórmula:
$$ valor = \frac {X-media}{desviación} $$
Ejercicio 3.C
Usando la función plot_metrics muestra las gráficas de las pérdidas por épocas de las siguientes redes neuronales para el DataFrame de las flores:
| Nº Neuronas en cada capa |
|---|
| 2,4,2,1 |
| 4,8,4,1 |
| 8,16,8,1 |
| 4,8,4,2,1 |
| 8,16,8,4,1 |
| 16,32,16,8,1 |
| 32,64,32,8,1 |
| 64,128,64,8,1 |
| 8,16,32,64,32,16,8,1 |
Pero para cada tipo de red ,deberás entrenarla 3 veces distintas y obtener resultado distintos con:
- Los datos originales
- Los datos normalizados
- Los datos estandarizados
Además:
- Deberás mostrar los 9 subplots en la disposición de 9x3 (9 filas y 3 columnas)
- En la primera columna se mostrarán el resultado de entrenar la red con los datos originales
- En la segunda columna se mostrarán el resultado de entrenar la red con los datos normalizados
- En la tercera columna se mostrarán el resultado de entrenar la red con los datos estandarizados
- El título de cada subplot:
- En la primera columna se mostrarán nº de neuronas por capa y el texto "originales"
- En la segunda columna se mostrarán nº de neuronas por capa y el texto "normalizados"
- En la tercera columna se mostrarán nº de neuronas por capa y el texto "estandarizados"
¿Hay diferencias al usar los datos normalizados o estandarizado? ¿Cual es mejor y peor? ¿Las ventajas son las mismas independientemente de la red?