Herramientas de usuario
−Barra lateral
Probabilidad métricas
Vamos a explicar como calcular la probabilidad que da una red neuronal en función de la prevalencia y el resultado de la red.
Para ello vamos a imaginar que la red neuronal, analiza una imágen para decirnos si tenemos cancer. El resultado puede ser positivo si tenemos cáncer y negativo si no tenemos cáncer.
- Negativo=0
- Positivo=1
Para calcular las métricas tenemos 2 arrays:
- Yreal={yreal∈N∣yreal∈{0,1}} Array con los valores reales o verdaderos.
- Ypredicho={ypredicho∈R∣ypredicho∈[0,1[} Array con los valores predichos por la red neuronal.
y ahora los juntamos en un único conjunto
- Y={(yreal,ypredicho)|yreal∈Yreal,ypredicho∈Ypredicho} Parejas de datos reales y predichos.
El umbral
Y vamos a suponer que el umbral para saber si es positivo o negativo es 0.5
N=|{(yreal,ypredicho)∈Y|yreal=0}|
P=|{(yreal,ypredicho)∈Y|yreal=1}|
PN=|{(yreal,ypredicho)∈Y|ypredicho∈[0,0.5[}|
PP=|{(yreal,ypredicho)∈Y|ypredicho∈[0.5,1[}|
TN=|{(yreal,ypredicho)∈Y|yreal=0∧ypredicho∈[0,0.5[}|
FP=|{(yreal,ypredicho)∈Y|yreal=0∧ypredicho∈[0.5,1[}|
FN=|{(yreal,ypredicho)∈Y|yreal=1∧ypredicho∈[0,0.5[}|
TP=|{(yreal,ypredicho)∈Y|yreal=1∧ypredicho∈[0.5,1[}|
La matriz de confusión es la siguiente:
| Predicción | |||
|---|---|---|---|
| PN,Negativo | PP,Positivo | ||
| Realidad | N,Negativo | TN | FP |
| P,Positivo | FN | TP | |
Sin embargo tanto la predicción como la realidad son números , por lo tanto se puede expresar así:
| Predicción | |||
|---|---|---|---|
| PN,ypredicho∈[0,0.5[ | PP,ypredicho∈[0.5,1[ | ||
| Realidad | N,yreal=0 | TN | FP |
| P,yreal=1 | FN | TP | |
Ahora mostremos las métricas
| Predicción | Métricas | ||||
|---|---|---|---|---|---|
| PN,ypredicho∈[0,0.5[ | PP,ypredicho∈[0.5,1[ | Predicho [0,0.5[ | Predicho [0.5,1[ | ||
| Realidad | N,yreal=0 | TN | FP | TNR=TNN | FPR=FPN |
| P,yreal=1 | FN | TP | FNR=FNP | TPR=TPP | |
| Métricas | Negativa | NPV=TNPN | FDR=FPPP | ||
| Positiva | FOR=FNPN | PPV=TPPP | |||
P(Predichonegativo|Negativo)=TNN=TrueNegativeRate(TNR)=EspecificidadP(Predichopositivo|Negativo)=FPN=FalsePositiveRate(FPR)=1−EspecificidadP(Predichonegativo|Positivo)=FNP=FalseNegativeRate(FNR)=1−SensibilidadP(Predichopositivo|Positivo)=TPP=TruePositiveRate(TPR)=Sensibilidad
P(Negativo|Predichonegativo)=TNPN=NegativePredictiveValue(NPV)P(Positivo|Predichonegativo)=FPPN=FalseOmissionRate(FOR)=1−NPVP(Negativo|Predichopositivo)=FPPP=FalseDiscoveryRate(FDR)=1−PrecisiónP(Positivo|Predichopositivo)=TPPP=PositivePredictiveValue(PPV)=Precisión
P(Positivo)=PP+N=PrevalenciaP(Negativo)=NP+N=1−PrevalenciaP(Predichopositivo)=PPP+NP(Predichonegativo)=PNP+N
Pero usando Bayes podemos calcular los valores predictivos positivos y negativos usando la prevalencia que queramos:
P(Positivo|Predichopositivo)=P(Predichopositivo|Positivo)∗P(Positivo)P(Predichopositivo|Positivo)∗P(Positivo)+P(Predichopositivo|Negativo)∗P(Negativo)
P(Positivo|Predichonegativo)=P(Predichonegativo|Positivo)∗P(Positivo)P(Predichonegativo|Positivo)∗P(Positivo)+P(Predichonegativo|Negativo)∗P(Negativo)
P(Negativo|Predichonegativo)=P(Predichonegativo|Negativo)∗P(Negativo)P(Predichonegativo|Negativo)∗P(Negativo)+P(Predichonegativo|Positivo)∗P(Positivo)
P(Negativo|Predichopositivo)=P(Predichopositivo|Negativo)∗P(Negativo)P(Predichopositivo|Negativo)∗P(Negativo)+P(Predichopositivo|Positivo)∗P(Positivo)
Simplificando las fórmulas
Ahora vamos a cambiar las fórmulas para en vez de poner por rangos de [0,0.5[ y [0.5,1[ usaremos un conjunto de rangos llamado I:
I={[0,0.5[,[0.5,1[}
Por lo tanto ahora habrá menos fórmulas.
N=|{(yreal,ypredicho)∈Y|yreal=0}|
P=|{(yreal,ypredicho)∈Y|yreal=1}|
Prei=|{(yreal,ypredicho)∈Y|ypredicho∈Ii}|
Ni=|{(yreal,ypredicho)∈Y|yreal=0∧ypredicho∈Ii}|
Pi=|{(yreal,ypredicho)∈Y|yreal=1∧ypredicho∈Ii}|
| Predicción | |||
|---|---|---|---|
| Pre0,ypredicho∈I0 | Pre1,ypredicho∈I1 | ||
| Realidad | N,yreal=0 | N0 | N1 |
| P,yreal=1 | P0 | P1 | |
P(Predichoi|Negativo)=NiNP(Predichoi|Positivo)=PiP
P(Negativo|Predichoi)=NiPreiP(Positivo|Predichoi)=PiPrei
P(Positivo)=PP+N=PrevalenciaP(Negativo)=NP+N=1−PrevalenciaP(Predichoi)=PreiP+N
Ahora mostremos las métricas
| Predicción | Métricas | ||||
|---|---|---|---|---|---|
| Pre0,ypredicho∈I0 | Pre1,ypredicho∈I1 | Predicho0 | Predicho1 | ||
| Realidad | N,yreal=0 | N0 | N1 | P(Predicho0|Negativo)=N0N | P(Predicho1|Negativo)=N1N |
| P,yreal=1 | P0 | P1 | P(Predicho0|Positivo)=P0P | P(Predicho1|Positivo)=P1P | |
| Métricas | Negativa | P(Negativo|Predicho0)=N0Pre0 | P(Negativo|Predicho1)=N1Pre1 | ||
| Positiva | P(Positivo|Predicho0)=P0Pre0 | P(Positivo|Predicho1)=P1Pre1 | |||
y ahora la tabla de confusión se podría simplificar aun más
| Predicción | Métricas | ||
|---|---|---|---|
| Prei,ypredicho∈Ii | Predichoi | ||
| Realidad | N,yreal=0 | Ni | P(Predichoi|Negativo)=NiN |
| P,yreal=1 | Pi | P(Predichoi|Positivo)=PiP | |
| Métricas | Negativa | P(Negativo|Predichoi)=NiPrei | |
| Positiva | P(Positivo|Predichoi)=PiPrei | ||
Volvemos a usar Bayes para calcular la precisión usando la prevalencia que queramos:
P(Positivo|Predichoi)=P(Predichoi|Positivo)∗P(Positivo)P(Predichoi|Positivo)∗P(Positivo)+P(Predichoi|Negativo)∗P(Negativo)
P(Negativo|Predichoi)=P(Predichoi|Negativo)∗P(Negativo)P(Predichoi|Negativo)∗P(Negativo)+P(Predichoi|Positivo)∗P(Positivo)=1−P(Positivo|Predichoi)
Generalizando los intervalos
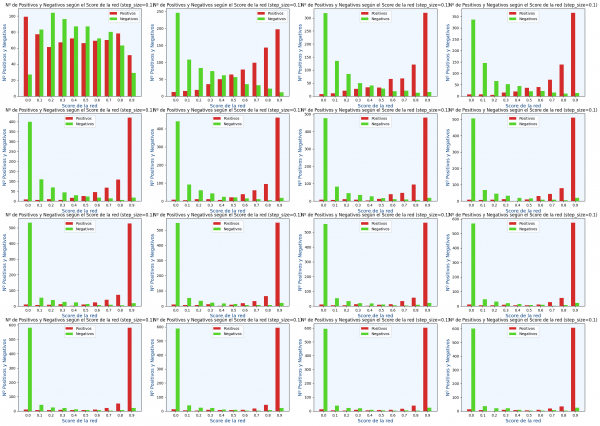
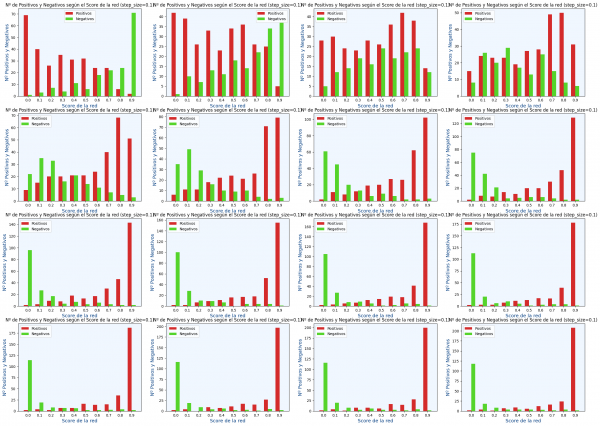
y ahora viene lo interesante, ¿que pasaría si en vez de haber un único umbral hubiera 10 umbrales?
I={[0,0.1[,[0.1,0.2[,[0.2,0.3[,[0.3,0.4[,[0.4,0.5[,[0.5,0.6[,[0.6,0.7[,[0.7,0.8[,[0.8,0.9[,[0.9,1[}
En el siguiente código en Python se puede ver el resultado de usar las fórmulas:main.ipynb
Infinitos intervalos
¿Y si hubiera infinitos intervalos? Que podríamos sacar la probabilidad exacta para el resultado que nos ha dado.
¿como sacarlo para infinitos intervalos? Pues calculando la función de densidad de probabilidad.
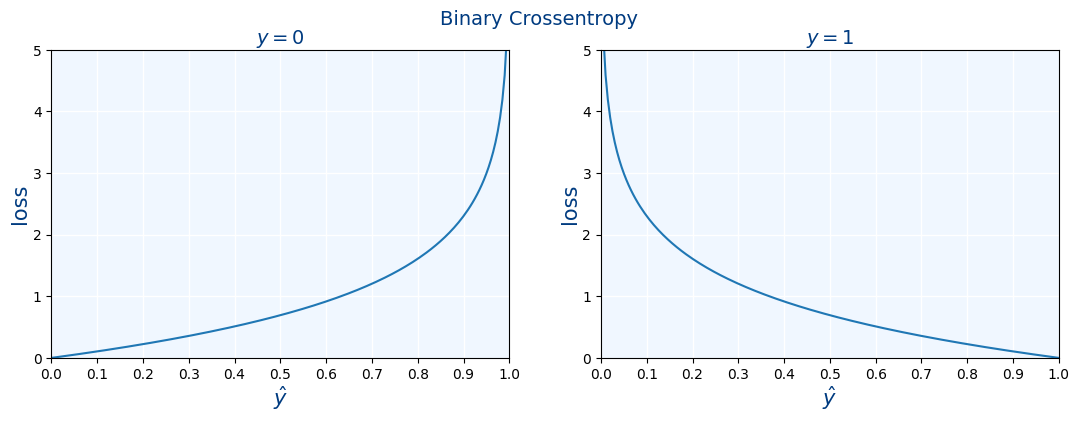
Sabemos que la función de coste para las redes neuronales es BinaryCrossEntropy:
BinaryCrossEntropy(yreal,ypredicho)=−(yreal⋅log(ypredicho)+(1−yreal)⋅log(1−ypredicho))
Los valores de yreal son 0 o 1, mientras que los valores de ypredicho es un número real entre 0 y 1
Veamos ahora gráficamente como es la fórmula según si yreal=0 o yreal=1
BinaryCrossEntropy(yreal,ypredicho)={−log(1−ypredicho)siyreal=0−log(ypredicho)siyreal=1